聯(lián)系電話
- 聯(lián)系人:
- 曹女士
- 電話:
- 400-6111-883
- 手機:
- 售后:
- 4006-111-883
- 傳真:
- 86-21-34615995
- 地址:
- 上海市浦東新區(qū)天雄路166弄1號3樓
- 網(wǎng)址:
- www.yeasen.com
掃一掃訪問手機商鋪
在探索生命科學(xué)的奧秘中,,紫杉醇這一神奇的藥物始終占據(jù)著舉足輕重的地位,。作為一種抗擊癌癥的利器,,紫杉醇的結(jié)構(gòu)復(fù)雜,它由一個高度活躍的碳骨架和一條帶有苯基異亮氨酸的側(cè)鏈構(gòu)成,。然而,,正是這種構(gòu)造,使得紫杉醇的合成之路充滿了挑戰(zhàn),。

研究人員對紫杉醇的生物合成路徑進(jìn)行了長達(dá)半個世紀(jì)的研究,,但仍有許多關(guān)鍵的轉(zhuǎn)化步驟隱藏在迷霧之中。特別是,,氧化四環(huán)烷環(huán)的形成和C9位碳原子的氧化過程,,至今仍是一個未解之謎,它們像兩座高峰,,阻擋在我們理解紫杉醇合成路徑的道路上,。
在這條合成路徑中,找到負(fù)責(zé)氧化四環(huán)烷環(huán)和C9位氧化的關(guān)鍵酶,,就像是找到了一把鑰匙,,能夠?qū)⒆仙即忌锖铣傻母鱾€孤立環(huán)節(jié)串聯(lián)起來,形成一條完整的鏈條,。
隨著紫杉醇在癌癥治療中的應(yīng)用越來越廣泛,,市場對它的需求也在不斷攀升。然而,,目前依賴天然資源提取的半合成生產(chǎn)方法,,已經(jīng)無法滿足這種日益增長的需求,。這就像一條狹窄的通道,無法承載日益增多的行人,。
幸運的是,,合成生物學(xué)的興起為我們提供了一條新的道路。通過合成生物學(xué),,我們有望實現(xiàn)紫杉醇的綠色,、可持續(xù)生產(chǎn)。因此,,揭開紫杉醇生物合成路徑的神秘面紗,,不僅能夠滿足市場需求,更能夠推動合成生物學(xué)的發(fā)展,,開啟一個全新的綠色制藥時代,。

2024年1月26日,國際頂級期刊Science(IF:56.9)在線發(fā)表了基因組所閆建斌研究員與北京大學(xué)雷曉光教授等合作完成的最新研究:“Characterization and heterologous reconstitution of Taxus biosynthetic enzymes leading to baccatin III",。該研究不僅揭示了紫杉醇生物合成中的關(guān)鍵酶和步驟,,而且為合成生物學(xué)生產(chǎn)紫杉醇提供了新的策略和方法,有助于減少對天然資源的依賴,,滿足市場對紫杉醇不斷增長的需求,。該研究成果標(biāo)志著我國在紫杉醇合成生物學(xué)理論和技術(shù)方面位于地位。
研究思路

環(huán)氧化催化關(guān)鍵酶-TOT1的解析
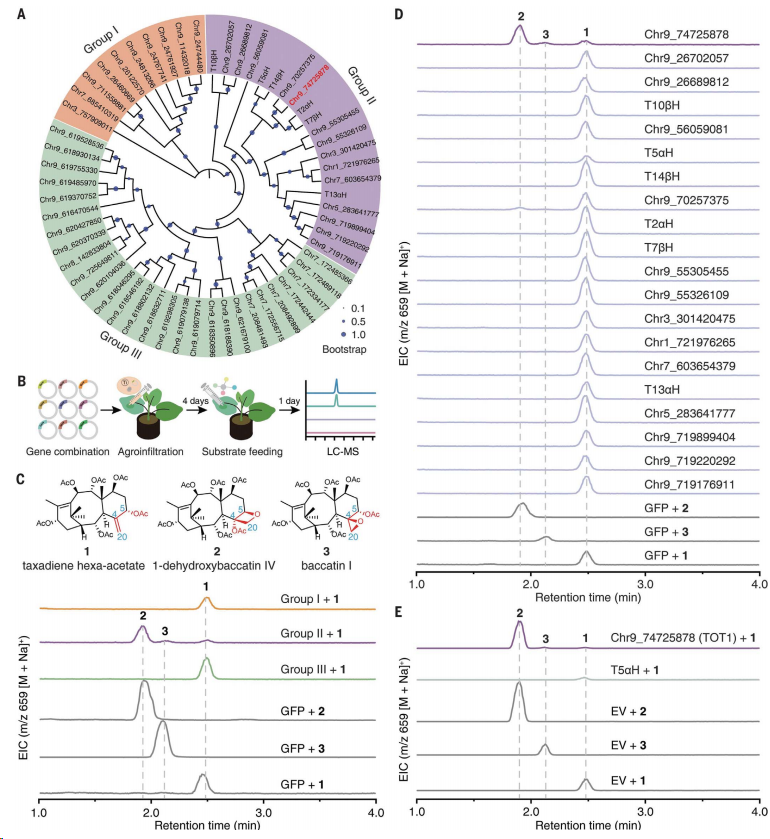
在探索紫杉醇這一神奇藥物的背后,,研究人員采取了一系列精妙的實驗步驟,,揭示了TOT1基因在氧化環(huán)形成中的關(guān)鍵角色。首先,,他們運用了一種巧妙的篩選策略,。基于紫杉烷生物合成途徑的知識,,研究人員將CYP725A家族的基因分為三個小組,,并在煙草葉片中同時表達(dá)了這三組基因。隨后,,他們使用液相色譜質(zhì)譜分析技術(shù)來檢測底物的氧化產(chǎn)物,。
接著,他們進(jìn)行了活性篩選,。在表達(dá)了第二組基因的葉片中,,研究人員觀察到了明顯的底物氧化產(chǎn)物峰,這表明該組基因中可能含有他們尋找的酶,。
為了驗證這一假設(shè),研究人員單獨表達(dá)了第二組中的每一個基因,。他們發(fā)現(xiàn),,當(dāng)Chr9_74725878基因被表達(dá)時,,底物氧化產(chǎn)物的數(shù)量顯著增加。這一發(fā)現(xiàn)證明了該基因編碼的蛋白質(zhì)具有催化氧化環(huán)形成的能力,。
基于這一發(fā)現(xiàn),,該基因被命名為TOT1,并通過體外表達(dá)和底物喂養(yǎng)實驗進(jìn)一步驗證了其功能,。
為了深入了解TOT1的作用機理,,研究人員運用了結(jié)構(gòu)模擬和反應(yīng)機理計算的方法。他們的研究揭示了TOT1介導(dǎo)的氧化環(huán)形成反應(yīng)機理,,即它能直接將烯基轉(zhuǎn)化為環(huán)氧化物和氧化環(huán),。
最后,為了驗證TOT1在紫杉醇生物合成中的關(guān)鍵作用,,研究人員在紫杉植物中敲低了TOT1的表達(dá),。他們觀察到,百菌素和紫杉醇的含量隨之降低,,這一結(jié)果進(jìn)一步證明了TOT1在紫杉醇生物合成中的重要性,。
(圖片來自原文)
紫杉醇氧化烷基環(huán)形成機制
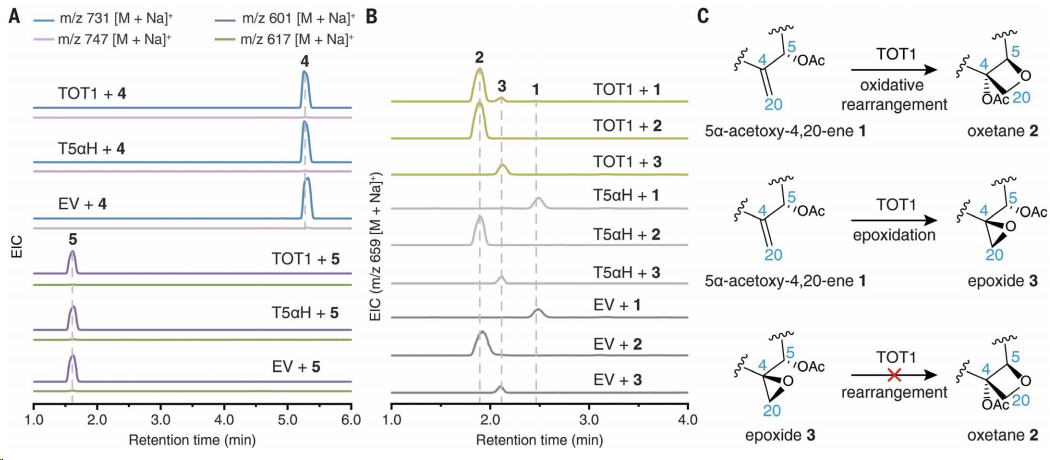
TOT1是一種具有雙重功效的氧化酶。能夠?qū)⒆仙即贾械南┗苯愚D(zhuǎn)化為環(huán)氧化物,,進(jìn)而形成氧化烷基環(huán),。這一過程無需經(jīng)過環(huán)氧化物作為中間體的傳統(tǒng)路徑,而是通過一種更為直接和高效的轉(zhuǎn)化方式,。
在TOT1的催化下,,烯基首先經(jīng)歷氧化反應(yīng),形成環(huán)氧化物,。然而,,這個環(huán)氧化物并非故事的終點,而是通過一系列結(jié)構(gòu)重排,,最終演變成為氧化烷基環(huán),。這一發(fā)現(xiàn)挑戰(zhàn)了人們對氧化烷基環(huán)形成機制的傳統(tǒng)認(rèn)知,為理解紫杉醇的生物合成過程帶來了新的視角,。
(圖片來自原文)
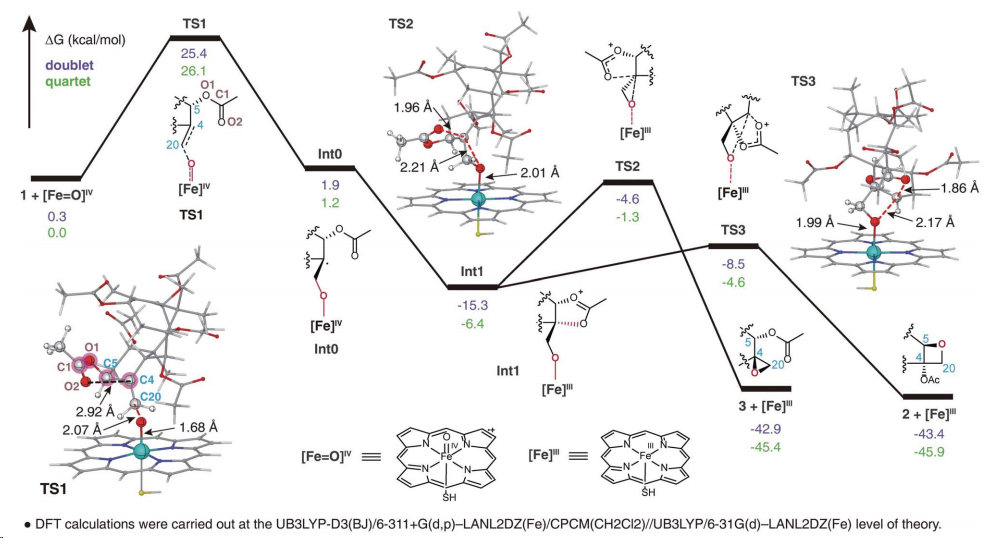
通過精確的計算模擬和深入的結(jié)構(gòu)分析,,研究人員揭示了TOT1催化氧化烷基環(huán)形成的詳細(xì)反應(yīng)機理。他們觀察到了過渡態(tài)和中間體的形成,,這些都是在TOT1催化下發(fā)生的微妙變化,。
(圖片來自原文)
特別值得注意的是,模擬計算結(jié)果顯示,,氧化烷基環(huán)產(chǎn)物的形成在能量上和動力學(xué)上都是優(yōu)先的,,這與實驗結(jié)果不謀而合。這一發(fā)現(xiàn)不僅驗證了計算模型的準(zhǔn)確性,,也為紫杉醇的生物合成提供了新的科學(xué)依據(jù),。
這一機制的創(chuàng)新之處在于,,它打破了我們以往對環(huán)氧化物作為中間體形成氧化烷基環(huán)的認(rèn)知壁壘,為氧化烷基環(huán)的形成提供了全新的解釋,。
C9羥基化酶的突破
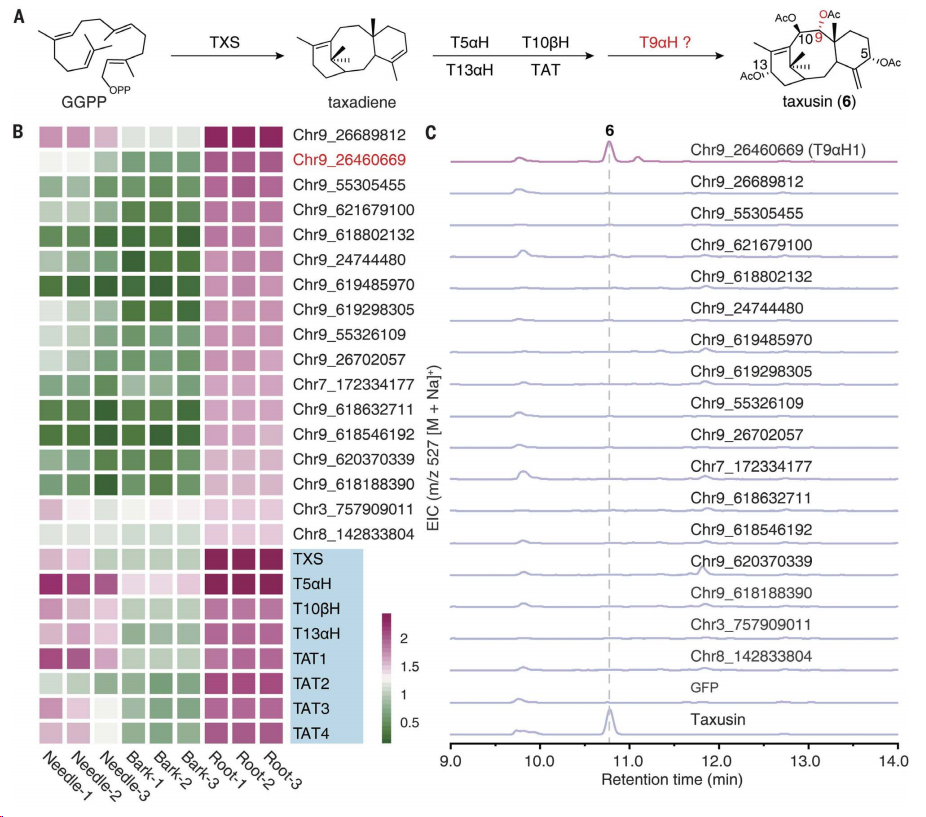
研究人員采取了一系列精確的實驗步驟,,最終揭示了關(guān)鍵的C9羥基化酶T9αH1。
首先,,研究人員對不同組織中的紫衫素含量進(jìn)行了分析,,發(fā)現(xiàn)紫衫素主要在Taxus植物的根部積累,而在針葉和樹皮中的含量則相對較低,。
接著,,他們對紫衫素生物合成途徑中已知基因的表達(dá)模式進(jìn)行了研究,發(fā)現(xiàn)這些基因在根部高度表達(dá),,與紫衫素的含量分布密切相關(guān),。
進(jìn)一步地,研究人員對CYP725A家族基因的組織特異性表達(dá)模式進(jìn)行了分析,,并從中篩選出了17個潛在的候選基因,。
為了鑒定這些候選基因中是否含有合成紫衫素所需的C9羥基化酶,研究人員在煙草植物中逐一表達(dá)了這些候選基因,,并同時表達(dá)了已知的紫衫素生物合成基因,。他們通過檢測是否能夠合成紫衫素來評估這些候選基因的功能。
實驗結(jié)果顯示,,只有Chr9_26460669基因的表達(dá)使得煙草能夠合成紫衫素,。這一發(fā)現(xiàn)證實了Chr9_26460669基因編碼了C9羥基化酶,并被命名為T9αH1,。
為了進(jìn)一步驗證T9αH1的C9羥基化酶活性,,研究人員在煙草中表達(dá)了T9αH1,并成功檢測到了紫衫素的生成,。
通過這一系列精妙的實驗步驟,,研究人員不僅成功從候選基因中篩選并鑒定出了C9羥基化酶T9αH1,而且補了巴卡丁III生物合成途徑中的一個關(guān)鍵空白,。
(圖片來自原文)
人工重建巴卡丁III生物合成途徑
研究人員首先在煙草葉片中同時共表達(dá)了已知參與巴卡丁III生物合成的基因,,包括TXS、T5αH,、T13αH,、T2αH、T7βH,、TAT和TBT,。
接著,他們在上述已知基因的基礎(chǔ)上,加入了新發(fā)現(xiàn)的兩個關(guān)鍵基因TOT和T9αH,,為巴卡丁III的生物合成途徑增添了新的力量,。
隨后,,研究人員在煙草葉片提取物中檢測到了一個新產(chǎn)物峰,,其質(zhì)譜和保留時間與巴卡丁III一致,這表明他們可能已經(jīng)成功合成巴卡丁III,。
為了進(jìn)一步確認(rèn)這個新產(chǎn)物的身份,,研究人員運用了MS/MS分析技術(shù),結(jié)果確認(rèn)該新產(chǎn)物確實是巴卡丁III,。
為了鑒定巴卡丁III生物合成的核心途徑,,研究人員逐一移除了每個基因,發(fā)現(xiàn)只有當(dāng)所有9個基因同時表達(dá)時才能產(chǎn)生巴卡丁III,,這證明了這9個基因構(gòu)成了巴卡丁III生物合成的核心途徑,。
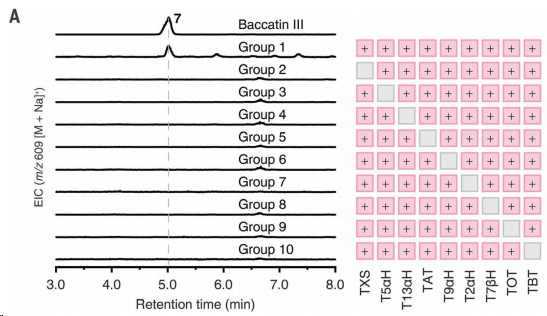
(圖片來自原文)
此外,他們還發(fā)現(xiàn)TAT不僅可以催化C10位的乙?;?,而且T10βH在C10位氧化反應(yīng)中可以被T5αH等其他酶取代。
特別值得注意的是,,盡管DBAT被認(rèn)為是C10位乙?;年P(guān)鍵酶,但研究人員發(fā)現(xiàn),,即使在沒有DBAT的情況下,,巴卡丁III仍然可以合成,這表明TAT可能也具有C10位的乙?;δ?。
通過人工共表達(dá)關(guān)鍵基因,研究人員成功地在煙草中重建了巴卡丁III的生物合成途徑,,為進(jìn)一步通過合成生物學(xué)生產(chǎn)紫杉醇提供了堅實的基礎(chǔ),。
核心基因的功能協(xié)同
在探索巴卡丁 III 生物合成過程的奧秘中,研究人員發(fā)現(xiàn)了一系列核心基因之間功能協(xié)同性的顯著特征,。首先,,TOT、T9αH等基因主要在根部表達(dá),,與下游基因的表達(dá)模式相似,,表明這些基因在組織水平上存在協(xié)同表達(dá)的現(xiàn)象。
其次,,經(jīng)過茉莉酸處理后,,這些基因的表達(dá)模式呈現(xiàn)協(xié)同上調(diào)的趨勢,與下游基因的表達(dá)模式相似,這提示茉莉酸可能參與協(xié)同調(diào)控這些基因,。
此外,,這些基因的表達(dá)之間存在顯著的正相關(guān)性,而與T14βH的表達(dá)模式不同,,這表明它們之間存在協(xié)同調(diào)控關(guān)系,。
在亞細(xì)胞水平上,這些酶主要定位于內(nèi)質(zhì)網(wǎng),,與質(zhì)體和細(xì)胞質(zhì)中的酶存在協(xié)同作用,,形成協(xié)同代謝途徑。
進(jìn)一步地,,這些基因構(gòu)成的代謝網(wǎng)絡(luò)呈現(xiàn)復(fù)雜的網(wǎng)絡(luò)狀而非線性,,形成協(xié)同的代謝途徑。
最后,,這些基因在染色體上的分布位置存在關(guān)聯(lián),,暗示它們之間可能存在協(xié)同進(jìn)化關(guān)系。
綜合來看,,這些核心基因在表達(dá)模式,、調(diào)控、亞細(xì)胞定位,、代謝途徑等方面存在協(xié)同性,,共同參與巴卡丁 III 的生物合成過程。
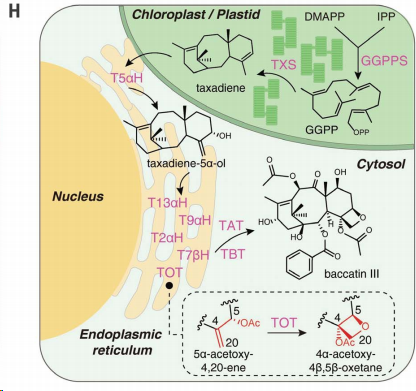
(圖片來自原文)
綜上所述,,本研究確定了紫杉科植物中催化生成抗癌藥物紫杉醇的關(guān)鍵酶,。紫杉醇的結(jié)構(gòu)復(fù)雜,包含一個功能化的二萜核心骨架(巴卡丁III)和苯異戊烯?;鶄?cè)鏈,。盡管在過去五十年中進(jìn)行了大量的研究,但巴卡丁III的完整生物合成途徑仍不清楚,。研究人員通過對紫杉醇生物合成途徑的篩選,,發(fā)現(xiàn)了一個雙功能細(xì)胞色素P450酶TOT1,它催化了紫杉醇中氧雜環(huán)的形成。通過共表達(dá)其他已知的參與巴卡丁 III合成的基因,,研究人員成功地在煙草中人工重建了巴卡丁 III的生物合成途徑,。這項研究為利用合成生物學(xué)手段綠色高效制造紫杉醇提供了可能。
翌圣助力產(chǎn)品
在該研究中,,研究團(tuán)隊使用了翌圣生物染料法熒光定量qPCR試劑進(jìn)行基因表達(dá)分析:

(圖片來自原文)
目前翌圣qPCR mix系列的產(chǎn)品已經(jīng)榮登Nature,、 Cell等多個頂級期刊,獲得科研大牛們認(rèn)可,!以下僅展示部分助力發(fā)表的高分文章:
[1] Li Y, Wang D, Ping X, et al. Local hyperthermia therapy induces browning of white fat and treats obesity. Cell. 2022;185(6):949-966.e19.doi:10.1016/j.cell.2022.02.004.(IF=66.850)
[2] Seki T, Yang Y, Sun X, et al. Brown-fat-mediated tumour suppression by cold-altered global metabolism. Nature. 2022;608(7922):421-428. doi:10.1038/s41586-022-05030-3. (IF=69.504)
[3] Chen P, Wang W, Liu R, et al. Olfactory sensory experience regulates gliomagenesis via neuronal IGF1. Nature. 2022;606(7914):550-556. doi:10.1038/s41586-022-04719-9.(IF=69.504)
[4] Dong W, Zhu Y, Chang H, et al. An SHR-SCR module specifies legume cortical cell fate to enable nodulation. Nature. 2021;589(7843):586-590. doi:10.1038/s41586-020-3016-z.(IF=69.504)
[5] Lu XY, Shi XJ, Hu A, et al. Feeding induces cholesterol biosynthesis via the mTORC1-USP20-HMGCR axis. Nature. 2020;588(7838):479-484. doi:10.1038/s41586-020-2928-y.(IF=69.504)
[6] Bi X, Wang K, Yang L, et al. Tracing the genetic footprints of vertebrate landing in non-teleost ray-finned fishes. Cell. 2021;184(5):1377-1391.e14. doi:10.1016/j.cell.2021.01.046.(IF=66.850)
[7] Liu S, Hua Y, Wang J, et al. RNA polymerase III is required for the repair of DNA double-strand breaks by homologous recombination. Cell. 2021;184(5):1314-1329.e10. doi:10.1016/j.cell.2021.01.048.(IF=66.850)
[8] Liu CX, Li X, Nan F, et al. Structure and Degradation of Circular RNAs Regulate PKR Activation in Innate Immunity. Cell. 2019;177(4):865-880.e21. doi:10.1016/j.cell.2019.03.046.(IF=66.850)
[9] Han X, Wang R, Zhou Y, et al. Mapping the Mouse Cell Atlas by Microwell-Seq. Cell. 2018;172(5):1091-1107.e17. doi:10.1016/j.cell.2018.02.001.(IF=66.850)
[10] Chai Q, Yu S, Zhong Y, et al. A bacterial phospholipid phosphatase inhibits host pyroptosis by hijacking ubiquitin. Science. 2022;378(6616):eabq0132. doi:10.1126/science.abq0132.(IF=63.714)
[11] Yu Q, Liu S, Yu L, et al. RNA demethylation increases the yield and biomass of rice and potato plants in field trials. Nat Biotechnol. 2021;39(12):1581-1588. doi:10.1038/s41587-021-00982-9.(IF=68.164)
[12] Han F, Liu X, Chen C, et al. Hypercholesterolemia risk-associated GPR146 is an orphan G-protein coupled receptor that regulates blood cholesterol levels in humans and mice. Cell Res. 2020;30(4):363-365. doi:10.1038/s41422-020-0303-z.(IF=46.297)
[13] Wang Z, Lu Z, Lin S, et al. Leucine-tRNA-synthase-2-expressing B cells contribute to colorectal cancer immunoevasion. Immunity. 2022;55(6):1067-1081.e8. doi:10.1016/j.immuni.2022.04.017.(IF=43.474)
[14] Bi Q, Wang C, Cheng G, et al. Microglia-derived PDGFB promotes neuronal potassium currents to suppress basal sympathetic tonicity and limit hypertension. Immunity. 2022;55(8):1466-1482.e9. doi:10.1016/j.immuni.2022.06.018.(IF=43.474)
[15] Wang X, Ni L, Wan S, et al. Febrile Temperature Critically Controls the Differentiation and Pathogenicity of T Helper 17 Cells. Immunity. 2020;52(2):328-341.e5. doi:10.1016/j.immuni.2020.01.006.(IF=43.474)
[16] Xiao J, Li W, Zheng X, et al. Targeting 7-Dehydrocholesterol Reductase Integrates Cholesterol Metabolism and IRF3 Activation to Eliminate Infection. Immunity. 2020;52(1):109-122.e6. doi:10.1016/j.immuni.2019.11.015.(IF=43.474)
[17] Zhang X, Zhang C, Qiao M, et al. Depletion of BATF in CAR-T cells enhances antitumor activity by inducing resistance against exhaustion and formation of central memory cells. Cancer Cell. 2022;40(11):1407-1422.e7. doi:10.1016/j.ccell.2022.09.013.(IF=38.585)
[18] Wang XY, Wei Y, Hu B, et al. c-Myc-driven glycolysis polarizes functional regulatory B cells that trigger pathogenic inflammatory responses. Signal Transduct Target Ther. 2022;7(1):105. Published 2022 Apr 18. doi:10.1038/s41392-022-00948-6.(IF=38.104)
[19] Fan H, Hong B, Luo Y, et al. The effect of whey protein on viral infection and replication of SARS-CoV-2 and pangolin coronavirus in vitro. Signal Transduct Target Ther. 2020;5(1):275. Published 2020 Nov 24. doi:10.1038/s41392-020-00408-z.(IF=38.104)
[20] Ren Y, Wang A, Wu D, et al. Dual inhibition of innate immunity and apoptosis by human cytomegalovirus protein UL37x1 enables efficient virus replication. Nat Microbiol. 2022;7(7):1041-1053. doi:10.1038/s41564-022-01136-6.(IF=30.964)
<上下滑動查看更多>
翌圣生物基因研究完整解決方案
產(chǎn)品特別推薦
方法 | 分類 | 產(chǎn)品名稱 | 貨號 |
RNA提取 | 同Trizol提取 | TRIeasy™ Total RNA Extraction Reagent | 10606ES |
免氯仿升級版 | TRIeasy™ Total RNA Extraction Reagent(Tcm Free) | 19202ES | |
動物組織/細(xì)胞總RNA提取,,避開有毒試劑,,最快15 min完成 | MolPure® Cell/Tissue Total RNA Kit細(xì)胞/組織總RNA提取試劑盒 | 19221ES | |
反轉(zhuǎn)錄試劑 | 5 min一步gDNA去除&反轉(zhuǎn)錄預(yù)混液(下游應(yīng)用qPCR) | Hifair® AdvanceFast One-step RT-gDNA Digestion SuperMix for qPCR | 11151ES |
5 min快速反轉(zhuǎn),最長可滿足14 kb cDNA合成,,含gDNA去除(下游應(yīng)用PCR/qPCR) | Hifair® AdvanceFast 1st Strand cDNA Synthesis Kit | 11149/11150ES | |
高質(zhì)量鏈cDNA合成預(yù)混液,,含gDNA去除(下游應(yīng)用qPCR) | Hifair® III 1st Strand cDNA Synthesis SuperMix for qPCR (gDNA digester plus) | 11141ES | |
qPCR染料法 | 高特異高熒光值定量預(yù)混液(染料法) | Hieff UNICON® Advanced qPCR SYBR Master Mix | 11185ES |
高靈敏通用型定量預(yù)混液(染料法) | Hieff UNICON® Universal Blue qPCR SYBR Master Mix | 11184ES | |
性價比定量預(yù)混液 (染料法),已發(fā)文章累計IF達(dá)到5000+ | Hieff® qPCR SYBR Green Master Mix (No Rox) | 11201ES | |
Hieff® qPCR SYBR Green Master Mix (Low Rox) | 11202ES | ||
Hieff® qPCR SYBR Green Master Mix (High Rox) | 11203ES |

 初級會員·13年
初級會員·13年
